|

|
|
|
11.2 Klassische Untersuchungsmethoden
|
|
|
Molekulare zytogenetische Methoden
|
|
|
|
Grosse Deletionen, Duplikationen, Inversionen und Translokationen, die DNA-Segmente von ca. 5-10 Millionen Basenpaare oder mehr umfassen können normalerweise nach konventioneller Bänderung der Chromosomen nachgewiesen werden. Dazu braucht es Chromosomen-Präparationen von guter Qualität mit gut gestreckten Chromosomen. Umfasst die Veränderung einen kleineren Abschnitt eines Chromosoms oder ist die Veränderung sehr komplex, reichen Bänderungsmuster der Chromosomen normalerweise nicht mehr aus, um eine exakte Beschreibung der Veränderung zu machen. Für solche Fälle müssen je nach der Fragestellung andere Techniken eingesetzt werden. Eine der wichtigsten Methoden ist die in situ Hybridisierung. Sie wird im Folgenden erklärt.
|
|
|
In Situ Hybridisierung (ISH)
|
|
|
|
Unterschiedliche Techniken der in situ Hybridisierung (ISH) können eingesetzt werden, um Gene zu kartieren und um einfache oder komplexe chromosomale Aberrationen besser charakterisieren zu können. Dazu werden oft BAC-Klone oder andere bekannte DNA-Sequenzen für spezifische Gene oder genetische Marker als DNA-Sonden eingesetzt.
Bevor die Hybridisierung durchgeführt wird, müssen die Chromosomen durch ein Bänderungsmuster identifiziert werden. Die Resultate (Fotografien) werden in einem Datenfile gespeichert, um später die Resultate der Hybridisierung korrekt einem Chromosomenpaar zuweisen zu können.
Damit eine Hybridisierung stattfinden kann, müssen die markierte DNA-Sonde und die DNA der Chromosomen (Target-DNA) vorgängig denaturiert werden, d.h. die komplementären Stränge müssen getrennt werden. Die auf dem Objektträger fixierten Chromosomen werden von RNA befreit und durch eine Formamid-Behandlung bei ca. 72°C denaturiert. Die DNA-Sonde wird direkt im Hybridisierungsbuffer, der ebenfalls Formamid enthält, bei derselben Temperatur denaturiert. Die denaturierte DNA-Sonde wird dann auf die Metaphasen-Chromosomen aufgetropft und das Ganze mit einem Deckglas und Rubber Cement versiegelt. Die Hybridisierung erfolgt in einer Feuchtekammer bei 37°C für mindestens 18 Stunden. Nach Ablauf der Inkubationszeit wird das Deckglas entfernt und die Metaphasen werden von nicht gebundenen Molekülen der DNA-Sonde befreit. Der Nachweis der markierten DNA-Sonde, die an die chromosomale DNA gebunden hat, kann direkt oder indirekt erfolgen. Dazu wird ein Fluoreszenz-Mikroskop mit verschiedenen Filtern benötigt. |
|
|
|
Heute werden ISH Experimente praktisch nur noch mit nicht-radioaktiv markierten DNA-Sonden durchgeführt und als Fluoreszenz in situ Hybridisierung (FISH) bezeichnet.
|
|
|
| Abb. 03 - Metaphase des Hausrindes |
|
Legende |
 |
|
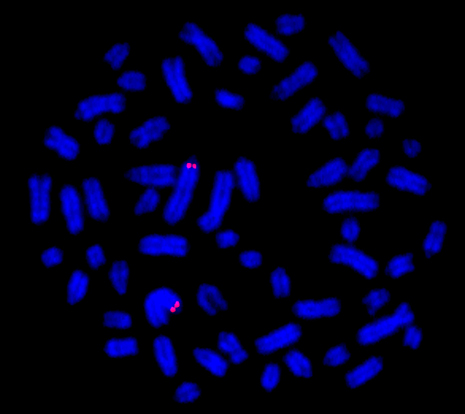
Abb. 03
Es sind klare und starke Signale nach Hybridisierung mit einer DNA-Sonde, die mit Texas Red markiert wurde, proximal auf dem Chromosom 1 (BTA 1) zu sehen. Diese DNA-Sonde hybridisiert in der Nähe des Genorts für Hornlosigkeit. Die Chromosomen wurden mit DAPI (blaue Färbung) angefärbt.
|
|
Eine Alternative zur Hybridisierung mit Metaphasen-Chromosomen ist die Hybridisierung auf Interphasen-Zellkernen. Dazu eignen sich beispielsweise Mundschleimhautabstriche sehr gut. Die Epithelzellen des Abstrichs werden auf einen Objektträger gebracht, fixiert und die DNA der Zellkerne denaturiert. Im Gegensatz zu den Metaphasen, ist die DNA der Interphasen-Zellkerne nicht kompaktiert. Trotzdem können die Signale (grün und pink) erkannt werden (siehe Abbildung 11 unten).
|
|
|
|
Für ein Double-FISH Experiment benötigen wir zwei DNA-Sonden, die vorgängig unterschiedlich markiert worden sind. Der Ablauf der Hybridisierung ist genau gleich wie bei einem normalen FISH-Experiment. Die DNA-Sonde kann dabei auf ein Einzelgen oder auf ein ganzes Chromosom gerichtet sein.
|
|
|
| Abb. 04 - Double FISH |
|
Legende |
 |
|
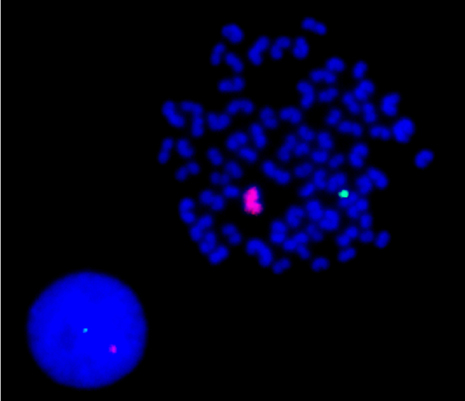
Abb. 04
Resultate eines Double-FISH Experiments für eine Metaphase (rechts oben) und für einen Interphase-Zellkern (links unten). Mit Texas Red und FITC (Fluorescein-isothiocyanat) markierte Chromosomen-Painting Proben decken das ganze X Chromosom bzw. Y Chromosom des Haushundes ab. In der Metaphase leuchten das X Chromosom und das Y Chromosom pink bzw. grün auf. Im Interphasenkern identische Situation.
|
|
Für eine FIBER-FISH (Abbildung 12) werden die Chromatinfäden (DNA) vor der Hybridisierung mechanisch künstlich gestreckt, indem man Gravitationskraft einwirken lässt. Damit kann die Auflösung der Analyse auf 1‘000 Basenpaare erhöht werden.
|
|
|
| Abb. 05 - FIBER-FISH Hybridisierungs-Experiments |
|
Legende |
 |
|
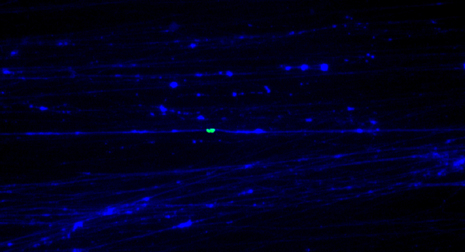
Abb. 05
In der Mitte sieht man eine von vielen Chromatin-Fibern. Grünes Signal zeigt eine bovine DNA-Sonde auf dem Rinderchromosom 3.
|
|
Eine Chromosomen-Painting-Probe ist ein Sonden-Gemisch von markierten DNA-Molekülen, die das gesamte Chromosom abdecken (siehe Abbildung 13). Um eine Chromosomen-Painting Probe herzustellen wird das Chromosom, für das man die Chromosomen-Painting Probe herstellen will, entweder durch ein sogenanntes Flow-Sorting-Verfahren oder nach Mikro-Dissektion von mehreren Kopien aus Metaphasen angereichert. Die so angereicherten DNA-Moleküle müssen mit Hilfe einer speziellen PCR (Degenerate-Oligonucleotide-PCR [=DOP-PCR]) angereichert und markiert werden. Chromosomen-Painting Proben erleichtern die Diagnostik. In Figur ist der Einsatz einer X Chromosomen-Painting Probe des Pferdes dargestellt.
|
|
|
| Abb. 06 - Equine X Chromosomen-Painting Probe für subfertile Stuten. |
|
Legende |
 |
|
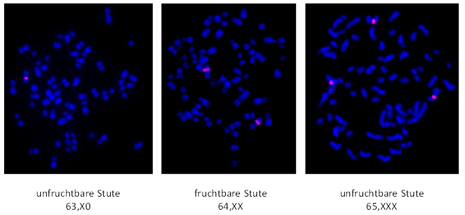
Abb. 06
Die X Chromosomen sind sehr leicht zu identifizieren.
|
|
Multicolor-FISH oder zu Deutsch "Vielfarb-Chromosomen-FISH" ist eine Technik, die zurzeit nur für wenige Arten (z.B. Mensch, Maus, Ratte) kommerziell verfügbar ist. Das Sonden-Gemisch enthält Chromosomen-Painting Proben für alle Chromosomen einer Spezies. Diese werden simultan hybridisiert und durch geschickte computergestützte Analysen können so unterschiedliche Farbimages für jedes homologe Chromosomenpaar erstellt werden. Diese Methode eignet sich sehr gut um einfache und komplexe strukturelle und numerische Chromosomen-Veränderungen nachzuweisen.
|
|
|
| Abb. 07 - Anwendung M-FISH für murine embryonale Stammzelle |
|
Legende |
 |
|
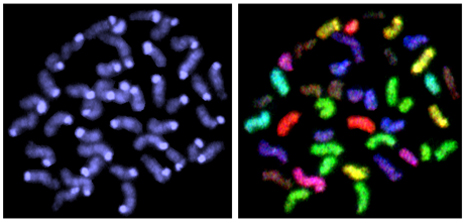
Abb. 07
Auf der linken Seite der Abbildung ist eine Metaphase aus murinen embryonalen Stammzellen zu sehen, die mit DAPI angefärbt wurde. Auf der rechten Seite ist dieselbe Metaphase, aber nach M-FISH-Experiment zu sehen. Es sind Aneuploidien und Trisomien sichtbar.
|
| Abb. 08 - Karyogramm der Metaphase aus Abbildung 07 (murine embryonale Stammzelle). |
|
Legende |
 |
|
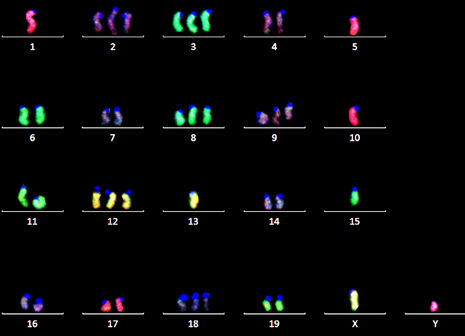
Abb. 08
Es sind Monosomien und Trisomien zu sehen.
|
Primed in situ Synthesis (PRINS)
|
|
|
|
Die Primed In Situ Synthesis (PRINS)-Methode beruht darauf, dass eine PCR direkt auf denaturierten Metaphasen-Chromosomen auf dem Objektträger stattfindet. Dazu sind spezielle PCR-Apparate entwickelt worden, in denen bis zu 16 Objektträger simultan eingesetzt werden können. Es können ein PCR-Primer oder zwei PCR-Primer (wie bei traditioneller PCR) eingesetzt werden.
|
|
|
| Abb. 09 - Anwendung PRINS für Telomere. |
|
Legende |
 |
|
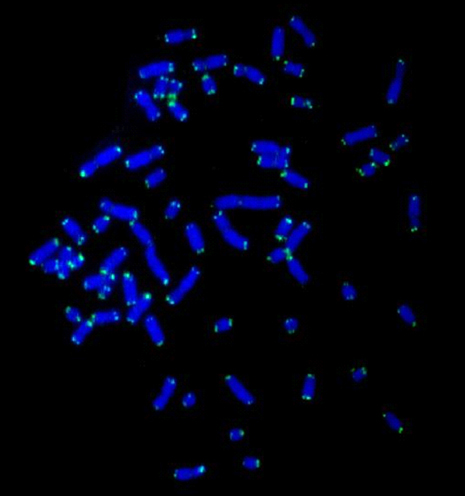
Abb. 09
In der Abbildung sind grüne Signale zu sehen. Diese Signale repräsentieren die durch PCR amplifizierten DNA-Sequenzen am Ende der Chromosomen.
|
Comparative genomic Hybridization (CGH)
|
|
|
|
Die Komparative genomische Hybridisierung (CGH) wird eingesetzt, um sogenannte Copy Number Variation (CNV) nachzuweisen. Dabei wird die genomische DNA eines Patienten grün markiert und zusammen mit einer rot markierten genomischen Referenz-DNA auf Metaphasen eines normalen männlichen Individuums hybridisiert. Computergestützte Farbimages zeigen Deletionen und Insertionen an. Eine Weiterentwicklung ist die sogennante micro-Array CGH.
|
|
|
|
|